大家好,这里是专注表观组学十余年,领跑多组学科研服务的易基因。
近日,中国农业大学玉米生物育种全国重点实验室董朝斌教授为第一及共同通讯作者,美国加州大学伯克利分校George Chuck研究员为共同通讯作者,在国际遗传学Top期刊《Nature Genetics》发表了题为“A regulatory network controlling developmental boundaries and meristem fates contributed to maize domestication”的研究论文。该研究鉴定出新的玉米驯化关键基因tasselsheath4(tsh4),并揭示tsh4位于传统玉米株型穗型驯化调控网络上游,阐明了驯化过程中玉米不同发育时期、不同性状的分子调控机理,为进一步开展玉米株型和穗型的遗传改良提供重要理论指导。

标题:A regulatory network controlling developmental boundaries and meristem fates contributed to maize domestication(发育边界和分生组织命运的调控网络促进玉米驯化)
发表时间:2024.10.16
发表期刊:Nature Genetics
影响因子:IF29/Q1
技术平台:ChIP-seq、GWAS、RNA-seq、miRNA-seq等(易基因金牌技术)
作者单位:中国农业大学董朝斌教授、美国加州大学伯克利分校George Chuck研究员、中国农业大学博士生胡高远、美国北卡州立大学陈秋月博士、美国加州大学伯克利分校Elena Shemyakina博士等。
DOI:10.1038/s41588-024-01943-z
在早期的驯化过程中,农民精心选育多种不同的营养和生殖生长性状的玉米,但由于这些性状存在异位显性(epistasis)和功能冗余性,因此其致病位点的鉴定一直未完全确定。本研究通过染色质免疫共沉淀测序(ChIP-seq)和全基因组关联分析(GWAS)的整合研究,鉴定出关键发育调控因子tasselsheath4(tsh4),参与调控营养生长和花序发育两阶段性状,且处于多个已知驯化位点的上游。TSH4作为新的玉米驯化因子,尽管其并不在分生组织中表达,但参与建立发育边界并决定分生组织命运。具体而言,TSH4通过双重负反馈机制发挥作用,该机制靶向抑制对其负调控的miRNA(microRNAs)。此外,TSH4与其同源基因存在功能冗余性,协同正调控一系列驯化相关位点,同时决定使现代玉米产量翻倍的分生组织。总之,TSH4在增产方面起关键作用,并助力形成理想的作物株型,从而成为驯化过程中的重要靶点。
易小结
本研究是表观遗传育种领域的一项标志性进展,系统性阐释了表观调控机制在作物驯化与改良中的核心作用,为分子设计育种提供了关键理论支撑与技术路径。该成果不仅凸显了表观遗传学在提升作物产量与环境适应性中的巨大潜力,也进一步拓展了表观基因组技术在农业育种中的应用前景。
易基因所提供的表观基因组学技术解决方案——包括ChIP-seq、m6A-seq和WGBS等——能够全面揭示基因表达调控网络,为指导精准育种和品种改良提供深入的科学依据。随着表观遗传学机制的持续解析,相关技术将在更多作物的遗传改良中发挥推动作用,助力农业向更高效、更智能、更可持续的方向发展。易基因相关产品拓展性案例展示
- 项目文章|Plant Physiol:郑州果树所王力荣团队ChIP-seq等揭示桃树需冷量和芽休眠调控的关键基因
- 项目文章|ChIP-seq及多组学技术揭示长期海洋变暖下硅藻的调控机制:资源分配权衡和表观遗传调控
- 项目文章|DNA甲基化与转录组联合分析助力揭示花青素生物合成调控柑橘果实品质的表观新机制
- 项目文章|Plant Cell Environ:WGBS+RNA-seq助力揭示植物不定根再生的DNA甲基化调控机制
- 项目文章|WGBS+RNA-seq揭示AtSAMS通过DNA甲基化和乙烯信号通路协同调控植物花器官发育的表观遗传机制
- PNAS:朱健康/郎曌博/牛庆丰团队ChIP-seq+WGBS揭示番茄果实成熟中DNA甲基化与转录因子互作的分子机制
- Cell封面:中国科学家杨学勇/黄三文m6A-seq等揭示同义突变通过表观转录调控机制决定生物性状|顶刊突破
研究方法
- 植物材料与生长条件:使用tsh4-mum1、ub2-mum1 和 ub3-mum1 突变体植株,通过回交和自交方法将这些突变体引入B73背景中。利用近等基因系(NILs)来比较研究玉米和野生玉米(teosinte)的tsh4等位基因。
- 全基因组关联分析(GWAS):利用866个玉米–野生玉米BC2S3重组自交系(RILs)和1257个BC1S4 RILs(TeoNAM群体),对多个驯化性状进行QTL-mapping,确定tsh4为候选基因。
- 染色质免疫沉淀测序(ChIP-seq):通过TSH4抗体对3-5mm的B73幼穗原基进行染色质免疫沉淀,鉴定TSH4的下游靶基因。
- 转录组分析(RNA-seq):对B73、tsh4、ub2/ub3和三重突变体的幼穗组织进行转录组测序,鉴定差异表达基因(DEGs)。
- 基因表达定量(RT-qPCR):通过RT-qPCR验证ChIP-seq和RNA-seq结果,分析tsh4及其靶基因的表达水平。
- 免疫定位(Immunolocalization):利用TSH4、UB2/UB3和TB1抗体对驯化玉米和野生玉米的幼穗和分生组织进行免疫定位分析。
- miRNA-seq:分析tsh4、ub2/ub3双突变体和三重突变体中的miRNA表达水平,鉴定TSH4靶向的miRNA。
- 基因多样性分析(Diversity Scans):对驯化玉米、地方品种玉米和野生玉米的tsh4基因进行核苷酸多样性分析。
结果图形
(1)驯化性状的全基因组关联分析
研究者对866个玉米–野生玉米BC2S3重组自交系和1,257个BC1S4 RILs(TeoNAM群体)进行了16个驯化性状的QTL-mapping。分析结果显示,多个与驯化相关的性状(如分蘖数、雄花序分支数、籽粒重量等)的QTL均定位到tsh4基因所在区间,表明tsh4是调控这些驯化性状的关键候选基因(图1a)。通过比较驯化玉米和野生玉米的tsh4等位基因,发现驯化玉米等位基因在减少分蘖数、雄花序分支数和提高籽粒重量方面具有显著效果(图1b-e)。这些结果表明,tsh4在玉米驯化过程中起到了重要作用。
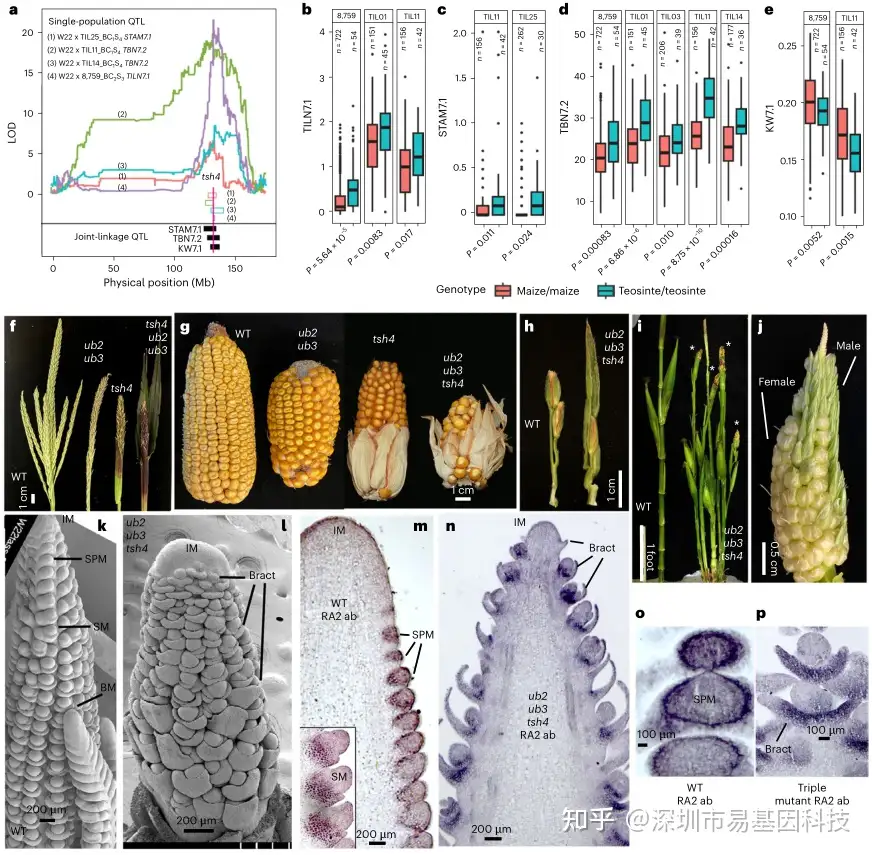
图1:tsh4影响驯化性状。
- 在六个不同的W22玉米-野生玉米比对群体中,四个驯化和改良的数量性状位点(QTLs)被定位到tsh4基因上。单群体QTL以显著LOD彩色曲线显示,多群体联合连锁TeoNAM QTLs显示在底部。QTL支持区间以水平条形图表示。tsh4基因位置由垂直红线标示。
b-e. 驯化玉米与野生玉米tsh4等位基因效应对比,野生玉米供体亲本列在顶部:(b) TILN7.1,(c) STAM7.1,(d) TBN7.2 和 (e) KW7.1。n = RILs的数量。
f. B73背景下的WT(野生型)、ub2-mum1/ub3-mum1、tsh4-mum1和tsh4-mum1/ub2-mum1/ub3-mum1三重突变体雄穗。在tsh4和三重突变体中移除了最下部苞叶,以揭示分支缺失。
g. WT、ub2-mum1/ub3-mum1、tsh4-mum1和三重突变体果穗。移除最下部苞叶以显示单籽粒。
h. 分解的WT野生型(左)和三重突变体苞叶腋中的单个小穗(右)。
- WT B73开花植株与三重突变体植株相比较。星号表示分蘖枝尖。
j. 三重突变体分蘖枝尖特写,显示混合性别特征。
k. WT雄穗的花序分生组织(IM)、小穗对分生组织(SPM)、小穗分生组织(SM)和分枝分生组织(BM)的SEM图像。
l. 去抑制的苞叶及BM或SPM缺失的三重突变体雄穗SEM图像。
m. 在WT中RA2的免疫定位显示在SPM中的表达。插图显示SM基部表达。
n. 在三重突变体中RA2的免疫定位显示在形成于分生组织之前的去抑制苞叶基部的异位表达。
o. WT SPM中RA2免疫定位的矢状视图,显示在基部由表达环形成的边界。
p. 三重突变体分生组织中RA2免疫定位的矢状视图,显示在苞叶中的异位表达和分生组织表达缺失。
(2)tsh4与ub2和ub3的功能冗余
研究发现,tsh4与其同源基因ub2和ub3在功能上存在冗余。通过分析ub2/ub3/tsh4三重突变体的雄花序和果穗,发现三重突变体表现出更严重的表型,包括雄花序分支缺失、小花序对缺失以及分蘖数增加(图1f-i)。这些表型在单突变体或双突变体中并未出现,表明tsh4、ub2和ub3在调控玉米发育过程中具有协同作用。
(3)TSH4的染色质免疫沉淀测序(ChIP-seq)
为鉴定TSH4的下游靶基因,研究者对3-5mm的B73幼穗原基进行了ChIP-seq分析。两个生物学重复共鉴定出2,609和3,136个高置信度的结合peaks,其中1,894个peaks在两个重复中均被检测到(图2a)。这些结合peaks大多位于基因区域(80.2%),且富集在GTAC核心结合位点(图2b-c)。通过与RNA-seq数据结合,研究者发现TSH4结合的基因中有263个在tsh4突变体中差异表达,这些基因主要参与转录调控、植物激素响应和发育过程(图2e)。
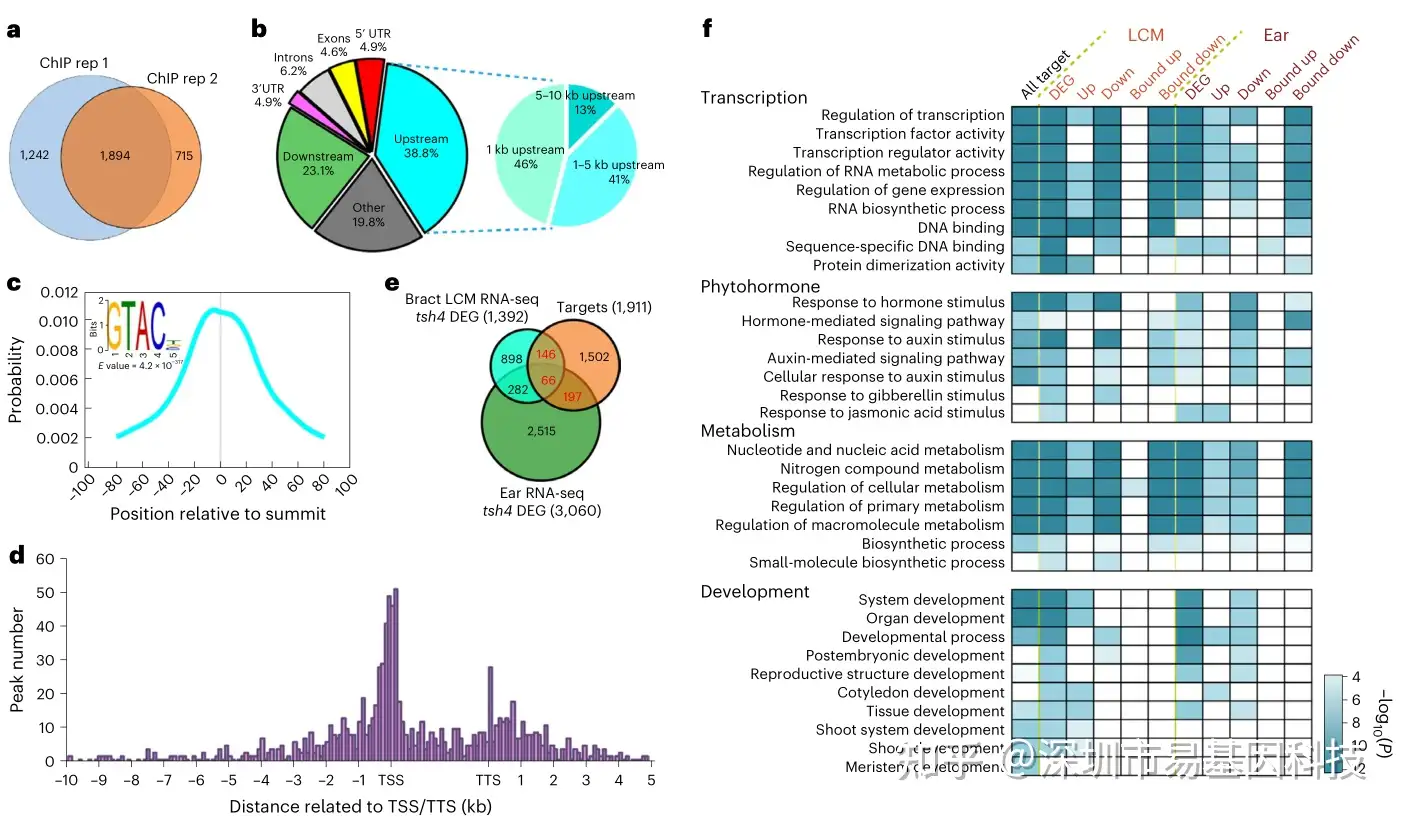
图2:利用ChIP-seq鉴定TSH4的直接靶标。
- 幼穗染色质相对于IgG对照的两个TSH4 ChIP–seq重复之间的重叠分析。
- 大多数peaks(超过80%)位于基因区域的全基因组TSH4结合peaks分布。
- TSH4结合peaks中GTAC SBP结合motifs的富集以及相对于peaks顶部位置。
- 相对于基因模型,TSH4结合peaks的分布。TSS,转录起始位点;TTS,转录终止位点。
- ChIP–seq鉴定的所有TSH4靶标与苞叶中LCM RNA-seq和幼穗组织的bulk RNA-seq鉴定的tsh4 DEGs进行重叠分析。
- 在e中鉴定的所有TSH4 ChIP–seq靶标和tsh4 DEGs的功能类别。
(4)TSH4靶向生长素响应调控因子
研究发现,TSH4能够结合并激活多个生长素响应基因家族成员,如Aux/IAA基因。这些基因在生长素信号中起负调控作用,其表达在tsh4、ub2/ub3双突变体和三重突变体中显著下调(图3c)。通过在三重突变体中引入生长素响应报告基因(DR5rev::mRFPer)和生长素转运报告基因(pZmPIN1a::ZmPIN1a-YFP),研究者发现三重突变体中生长素响应和转运主要发生在去抑制苞叶中,而野生型中则主要发生在分生组织中(图3d-e)。这表明TSH4通过调控生长素响应基因,影响苞叶和分生组织的发育。
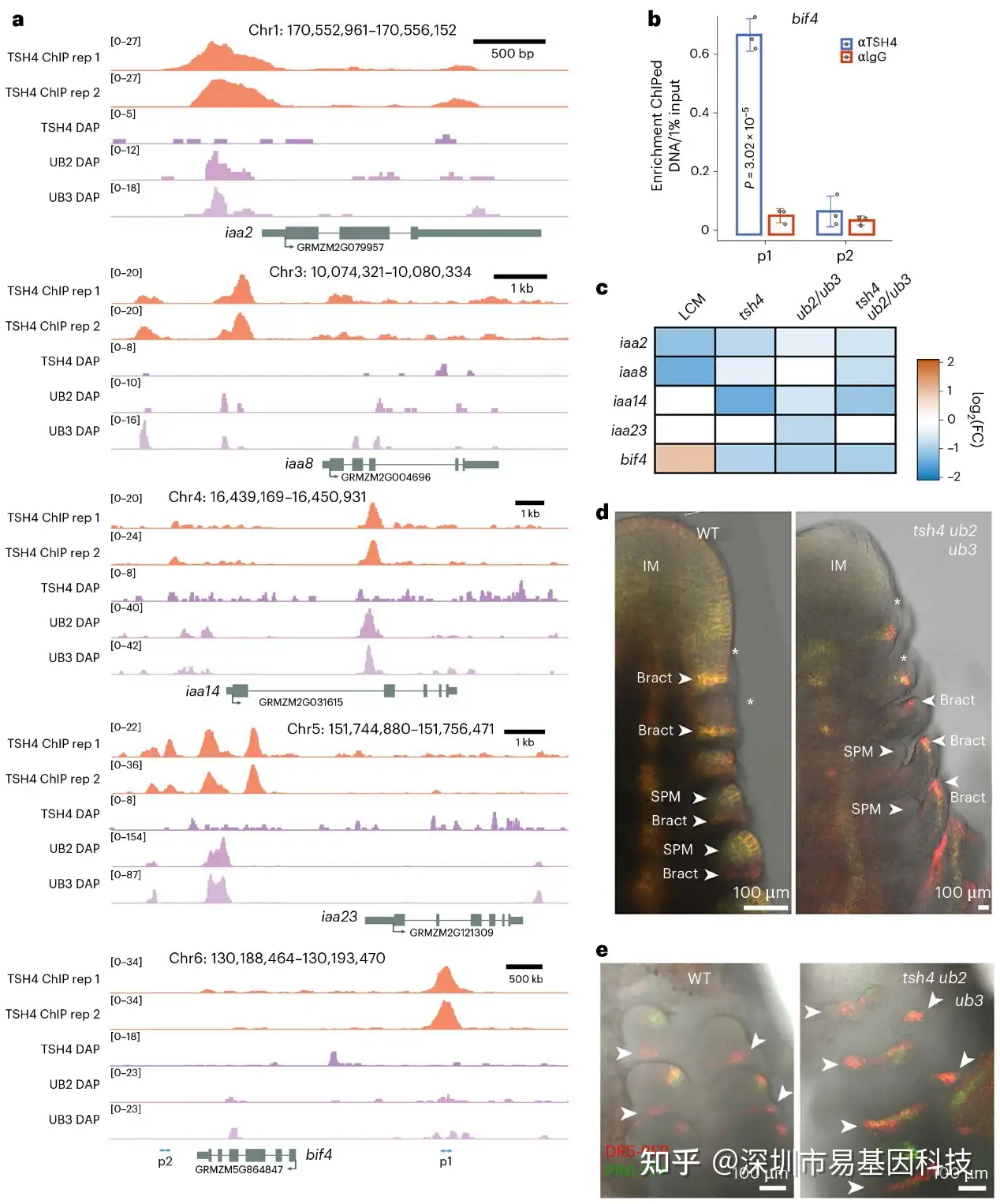
图3:TSH4、UB2和UB3调控生长素响应。
- 两个TSH4 ChIP–seq重复中Aux/IAA基因邻近的TSH4结合谱和TSH4、UB2和UB3 DAP-seq peaks。
- ChIP–qPCR(n=3个生物重复)验证TSH4 bif4结合peaks。与p2位点相对于IgG对照(橙色条)相比,位于ChIP–seq peaks的p1扩增区域在野生型(WT)染色质中显著富集(蓝色条)。
- 在tsh4苞叶、tsh4和ub2/ub3突变体以及三重突变体的果穗中Aux/IAA靶基因表达。
- 在WT(左)与三重突变体(右)果穗中的抑制苞叶和SPM生长素响应(Dr5rev::mRFPer)和转运(pZmPIN1a::ZmPIN1a-YFP)。
- WT(左)和三重突变体(右)SPM的矢状视图,显示苞叶(箭头)中的生长素响应和转运。
(5)TSH4靶向其自身的负调控miRNAs
研究发现,TSH4能够结合并抑制多个miRNA基因,包括MIR156和MIR529,这些miRNA能够降解tsh4 mRNA(图4a-b)。通过miRNA-seq分析,研究者发现这些miRNA在tsh4、ub2/ub3双突变体和三重突变体中表达上调(图4c)。通过同时进行miRNA原位杂交和TSH4免疫定位,研究者发现TSH4和MIR529在发育过程中逐渐建立互斥表达域,从而在分生组织和苞叶之间形成清晰的边界(图4e-g)。这种双重负反馈机制可能是TSH4调控发育边界的关键机制。
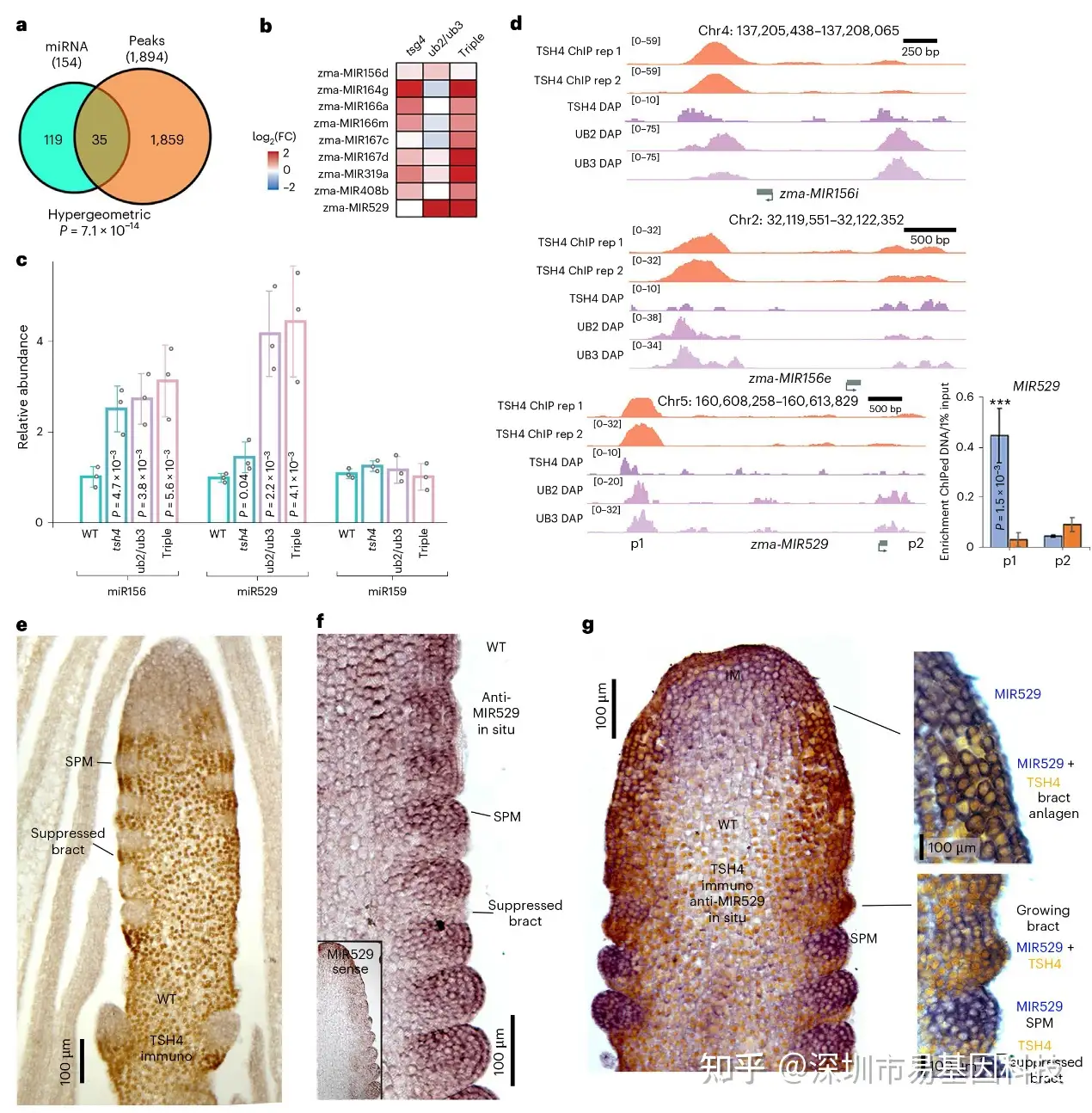
图4:TSH4与MIR529之间的双重负反馈调控。
- miRNA位点与TSH4结合peaks之间的重叠。
- miRNA-seq鉴定出在tsh4、双重突变体和三重突变体中上调的9个miRNA。
- RT-qPCR检测成熟miR156和miR529表达,显示在sbp突变体中上调。非靶标miR159没有差异。
- 代表性miRNA基因附近的TSH4结合谱示例,包括两个TSH4 ChIP–seq重复以及UB2、UB3和TSH4 DAP-seq peaks。插图显示位于peak(p1)和下游(p2)的引物对MIR529 peak的qPCR验证。
- 在幼穗上的TSH4免疫定位(金色)显示蛋白在茎、抑制苞叶中,但在IM和SPM中缺失。
- 反义MIR529原位杂交显示雄穗在SPM中表达,但在苞叶中不表达。插图显示了正义对照。
- MIR529反义miRNA原位(蓝色)和TSH4抗体(金色)对穗原基的双重标记。右侧放大插图在差分干涉对比(DIC)滤镜下拍摄,显示在苞叶原基(右上角)中的重叠,但在苞叶抑制阶段(右下角)重叠缺失和互补表达。
(6)TSH4靶向驯化位点
研究发现,TSH4能够结合并激活多个已知关键驯化基因,如tb1、tru1和tga1(图5a-c)。这些基因在玉米驯化过程中起到了关键作用,例如tb1调控分蘖抑制,tru1调控侧枝性别决定,tga1调控颖壳硬度。通过比较驯化玉米和野生玉米的tsh4等位基因,研究者发现驯化玉米等位基因在激活这些驯化基因方面更为高效(图5e)。这表明tsh4在玉米驯化过程中通过调控多个关键基因,促进了理想植物形态形成。
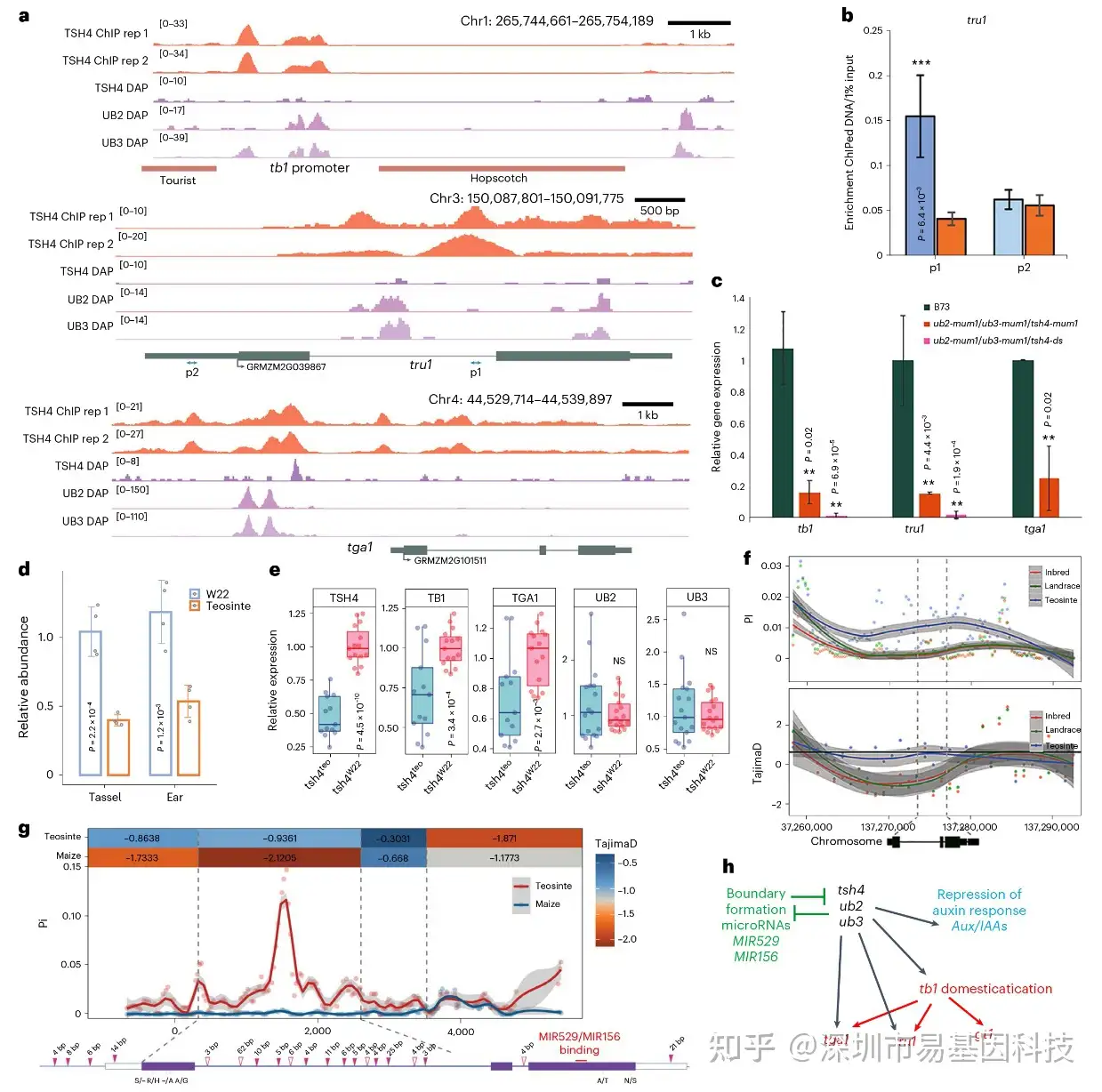
图5:TSH4靶向驯化位点。
- SBP结合peak位于tb1启动子上游58kb处,该位置紧邻负责驯化的Hopscotch反转录转座子插入区、tru1内含子区和tga1启动子区。
- ChIP–qPCR验证显示,与启动子(p2)相比,TSH4结合peak富集在tru1内含子(p1)中。
- 对3周龄B73和三重突变体(tb1和tru1)的幼苗以及吐丝期的果穗(tga1)中驯化基因表达的RT–qPCR分析。
- RT–qPCR比较驯化玉米tsh4与野生玉米tsh4在雄穗和果穗中的表达水平。
- 对驯化玉米/野生玉米NILs果穗组织的RT–qPCR显示,驯化玉米tsh4等位基因的品系(粉色)中tsh4、tb1和tga1上调。
- 驯化玉米、玉米地方品种和野生玉米中围绕tsh4位点区域的核苷酸多样性(PI)(上)和Tajima’sD检验(下)。垂直灰色虚线表示tsh4转录起始和终止位点。
- 15个野生玉米品系(红色)和25个玉米NAM群体自交系(蓝色)的序列数据评估tsh4基因内的核苷酸多样性(Pi)和Tajima’sD检验(顶部,彩色框)。tsh4的第一个内含子显示出最强的选择信号。下面是tsh4基因模型,显示相关多态性位点。实心箭头表示玉米等位基因相对于野生玉米插入,空心箭头表示玉米等位基因缺失;氨基酸变化标记在下方。
- 整合miRNA、生长素响应和驯化的tsh4功能模型。
(7)驯化玉米与野生玉米tsh4等位基因的活性差异
研究发现,驯化玉米tsh4等位基因在表达水平上高于野生玉米等位基因,且在激活驯化基因方面更为高效(图5d-e)。通过对15个野生玉米品系和25个驯化玉米NAM群体的核苷酸多样性分析,研究者发现tsh4基因在玉米驯化过程中受到强烈的正选择(图5f-g)。这些结果表明,高表达的玉米tsh4等位基因可能是玉米驯化过程中获得理想植物形态的关键因子。
结论和启示
本研究通过结合ChIP-seq、GWAS、RNA-seq和miRNA-seq等多种技术手段,揭示了tsh4在玉米驯化过程中的关键作用。tsh4通过调控多个驯化基因和miRNA,建立了发育边界,决定了分生组织命运,并显著提高了玉米产量。
ChIP-seq技术在本研究中发挥了重要作用,通过鉴定TSH4的下游靶基因,揭示了tsh4在调控玉米发育和驯化过程中的分子机制。未来类似研究可以利用ChIP-seq技术,结合其他组学技术,深入解析植物驯化和发育过程中的复杂调控网络,为作物改良提供新的理论依据。
关于易基因染色质免疫共沉淀测序 (ChIP-seq)
染色质免疫共沉淀(Chromatin Immunoprecipitation,ChIP),是研究体内蛋白质与DNA相互作用的经典方法。将ChIP与高通量测序技术相结合的ChIP-Seq技术,可在全基因组范围对特定蛋白的DNA结合位点进行高效而准确的筛选与鉴定,为研究的深入开展打下基础。
DNA与蛋白质的相互作用与基因的转录、染色质的空间构型和构象密切相关。运用组蛋白特定修饰的特异性抗体或DNA结合蛋白或转录因子特异性抗体富集与其结合的DNA片段,并进行纯化和文库构建,然后进行高通量测序,通过将获得的数据与参考基因组精确比对,研究人员可获得全基因组范围内某种修饰类型的特定组蛋白或转录因子与基因组DNA序列之间的关系,也可对多个样品进行差异比较。
应用方向:
ChIP 用来在空间上和时间上不同蛋白沿基因或基因组定位
- 转录因子和辅因子结合作用
- 复制因子和 DNA 修复蛋白
- 组蛋白修饰和变异组蛋白
技术优势:
- 物种范围广:细胞、动物组织、植物组织、细菌微生物多物种富集经验;
- 微量建库:只需5ng以上免疫沉淀后的DNA,即可展开测序分析;
- 方案灵活:根据不同的项目需求,选择不同的组蛋白修饰特异性抗体。
技术路线:
参考文献:
Dong Z, Hu G, Chen Q, Shemyakina EA, Chau G, Whipple CJ, Fletcher JC, Chuck G. A regulatory network controlling developmental boundaries and meristem fates contributed to maize domestication. Nat Genet. 2024 Oct 16. doi: 10.1038/s41588-024-01943-z.
相关阅读:
1.项目文章 | Nature子刊:ChIP-seq等揭示c-di-AMP与DasR互作以调控细菌生长、发育和抗生素合成
2. 项目文章 | Nat Commun:中南大学曾朝阳/熊炜/龚朝建团队利用ChIP-seq等揭示头颈鳞癌免疫逃逸机制
3. 项目文章 | ChIP-seq等揭示糖皮质激素和TET2共调控促进癌症转移的表观遗传机制
4. 项目文章:ChIP-seq等揭示人畜共患寄生虫弓形虫的蛋白质乳酸化和代谢调控机制
5. 项目集锦 | 易基因近期染色质免疫共沉淀测序(ChIP-seq)研究成果
6. 项目文章 | NAR:ChIP-seq等揭示蛋白质酰基化与c-di-GMP协同调控放线菌发育与抗生素合成机制
7. 项目文章|Plant Physiol:郑州果树所王力荣团队ChIP-seq等揭示桃树需冷量和芽休眠调控的关键基因
8.项目文章|CDD:ChIP+RNA-seq技术揭示NURR1在前列腺癌从基因转录到肿瘤进展中的调控机制
9.项目文章|mBio:ChIP-seq助力揭示AcP依赖性乙酰化修饰网络对放线菌c-di-AMP稳态的调控机制
10.项目文章|Sci Adv:WGBS+ChIP-seq揭示DNA甲基化在异基因造血干细胞移植后延迟血小板恢复中的关键作用
11. DNA与蛋白质互作及染色质开放性研究方案
12.项目文章|ChIP-seq及多组学技术揭示长期海洋变暖下硅藻的调控机制:资源分配权衡和表观遗传调控
13.项目文章|Hepatology/IF15.8:复旦中山医院沈英皓利用ChIP-seq及多组学解析肝癌仑伐替尼耐药机制(国人佳作)
14. 技术推介 | 染色质免疫共沉淀测序 (ChIP-seq)
农业生态专题
